De la Fuente y su equipo descubren con IA miles de antibióticos en organismos extintos
El científico gallego en EEUU lidera un trabajo que rescata moléculas de mamuts y otros animales desaparecidos hace miles de años

R. V.
Nuevo trabajo del grupo científico liderado por el científico gallego César de la Fuente en la Universidad de Pensilvania. Si hace apenas seis días anunciaba el descubrimiento de casi un millón de nuevos antibióticos en el microbioma global, en el cuerpo humano, ahora son los organismos extintos los explorados en busca de una solución a las bacterias resistentes. Y otra vez con la ayuda de la inteligencia artificial. La investigación se publica hoy en la revista “Nature Biomedical Engineering”.
El trabajo, titulado “Deep-learning-enabled antibiotic discovery through molecular de-extinction”, culmina el planteamiento que De la Fuente y su equipo, el Machine Biology Group, presentó en una preimpresión —trabajo no revisado por pares— en noviembre de 2022, la “desextinción molecular”.
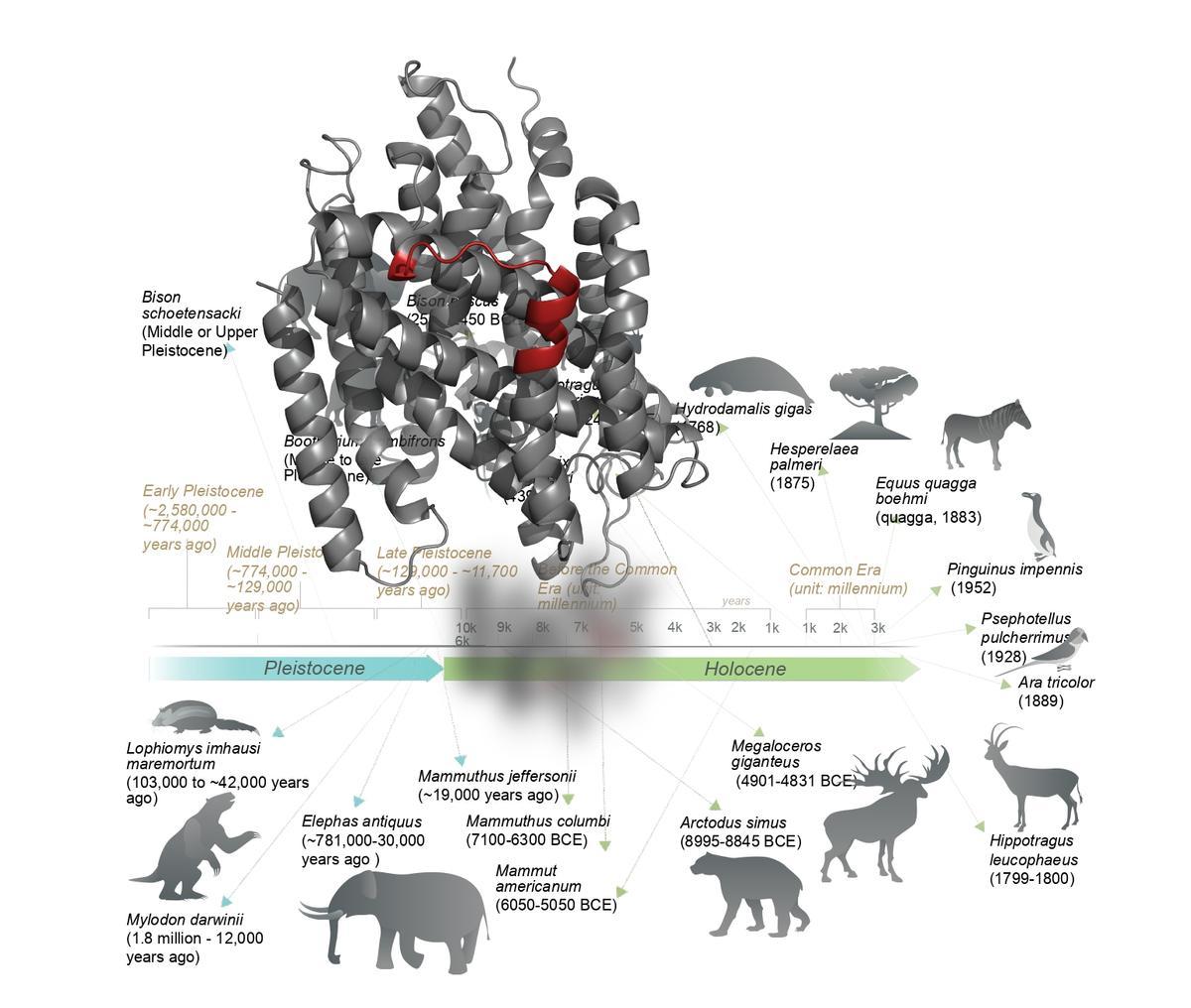
Se trata, por así decirlo, de la “resurrección” de moléculas de vida extintas: ácidos nucleicos, proteínas y otros compuestos que ya no están codificados por organismos vivos, y que se encontraban en humanos prehistóricos, como los neandertales, y animales extintos como el mamut lanudo y el milodón, una especie de perezoso gigante.
Estructura molecular / Cedida
Son solo tres ejemplos, porque los científicos liderados por el investigador gallego han explorado —aseguran— todos los organismos extintos conocidos por la ciencia, un logro que solo ha sido posible gracias a décadas de investigación previa en el desarrollo de métodos de secuenciación para material genético antiguo.
Este conjunto de genomas de organismos extintos, el “extintoma”, fue escudriñado mediante un modelo de aprendizaje profundo (“deep learning”) llamado sistema APEX, que ha logrado resucitar con éxito numerosos compuestos antibióticos encontrados en criaturas extintas hace miles de años.
El desarrollo de nuevos antibióticos mediante métodos tradicionales cuesta miles de millones de euros, tanto como una misión espacial, y puede prolongarse hasta 6 años para lograr candidatos preclínicos. Para acelerar el descubrimiento de nuevos antibióticos, el laboratorio de César de la Fuente creó un nuevo modelo de inteligencia artificial (IA) que permite descubrir cientos de miles de candidatos preclínicos en apenas unas horas.
Según informa el laboratorio de De la Fuente, muchos de los compuestos fueron efectivos tanto “in vitro” como en dos modelos preclínicos diferentes de ratón. La actividad de los compuestos principales fue comparable a la del antibiótico estándar de atención, la polimixina B, un grupo de antibióticos descubiertos en 1947 y que se utiliza para diversas indicaciones, como infecciones del tracto urinario y de la sangre, conjuntivitis y meningitis.

Recreación de un ejemplar de mamut lanudo con su “extintoma”. / Cedida
Las moléculas descubiertas han sido bautizadas con denominaciones como la neandertalina, la mamutina, la milodonina, y la megalocerina. La nendartalina procede de neandertales, mientras que la mamutina debe su nombre al mamut lanudo (Mammuthus primigenius), cuyo último remanente conocido sobrevivió en la isla de Wrangel, en el Ártico, hasta aproximadamente el año 1700 a. C. La milodonina, por su parte, se obtuvo de restos del milodón (Mylodon darwini), una especie extinta de mamífero emparentado con los actuales osos hormigueros y perezosos, aunque era de mucho mayor tamaño. Se extinguió hace unos 10.000 años y fue descubierta por Charles Darwin, de ahí su nombre científico. La megalocerina proviene del megalocero (Megaloceros giganteus), un ciervo gigante cuyas astas medían hasta 3,5 metros de punta a punta y que se extinguió hace unos 5.000 años.
Se han rastreado también especies vegetales y animales desaparecidos más recientemente, como el perico del paraíso (Psephotellus pulcherrimus), un ave endémica del este de Australia que se declaró extinta en 1928.
La crisis mundial de resistencia a los antibióticos, la amenaza de patógenos emergentes y el uso excesivo de antibióticos tradicionales requieren nuevos paradigmas de desarrollo de fármacos asistidos por herramientas informáticas. Los resultados obtenidos en esta especie de “farmacia prehistórica” abren nuevas perspectivas para el descubrimiento de antibióticos en información biológica, como genomas y proteomas, usando algoritmos de inteligencia artificial. Este avance es importante, ya que se estima que las bacterias resistentes a los antibióticos matarán a 10 millones de personas al año de aquí a 25 años.
César de la Fuente Núñez (A Coruña, 1986), que hace unos días ingresó como académico más joven en la Real Academia de Farmacia de Galicia, es profesor catedrático en la Universidad de Pensilvania (Estados Unidos). Licenciado en Biotecnología por la Universidad de León y doctor en microbiología e immunología por la Universidad de la Columbia Británica (Canadá), ha realizado estancias de investigación y formación en destacados centros como el Instituto de Tecnología de Massachusetts (MIT, Estados Unidos), y en 2019 fue reconocido por el “MIT Technology Review” como uno de los innovadores más importantes del mundo por “digitalizar la evolución para crear antibióticos mejores”. En 2021 recibió el Premio Princesa de Girona de Investigación Científica.
- Máxima alerta de la Policía Nacional por el mensaje que está llegando a los móviles de miles de personas
- Una belleza de armas tomar
- Un nutricionista desvela qué es lo primero que le quita a los pacientes que quieren adelgazar
- Uno de los dos tripulantes gallegos desaparecidos sobrevivió al secuestro del atunero Alakrana del 2009
- El abandono no entiende de razas ni de dinero: mascotas que valen miles de euros llegan a A Madroa
- Condenan a dos años y medio de prisión al maquinista del Alvia y al exdirector de Seguridad de ADIF por el accidente de Angrois
- La familia del vigués que murió tras el concierto de Karol G anuncia denuncia: 'No hubo forcejeo ni pelea
- Mujer de más de 45 y sola busca ayuda
